Modelado molecular en Linux
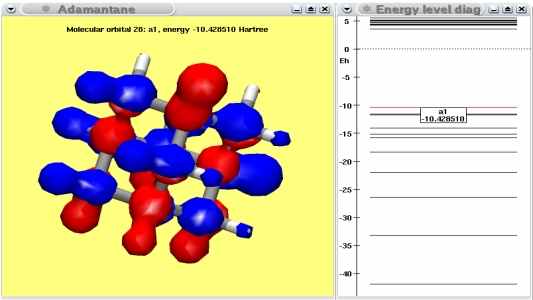
Los programas para modelado molecular pretenden simular, explicar o predecir la estructura tridimensional y las propiedades fisicoquímicas de las moléculas.
Equivalencias en Windows: Millsian (millsian.com), AgileMolecule (agilemolecule.com).
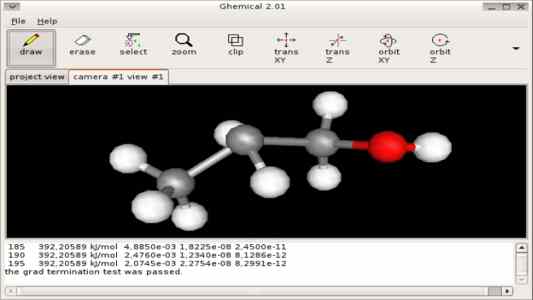
- Ghemical (bioinformatics.org/ghemical, paquete ghemical): programa para 'modelado molecular' integrado con OpenBabel (conversor de formatos de archivo químicos).
- Viewmol (viewmol.sourceforge.net, paquete viewmol): programa para 'modelado molecular'.
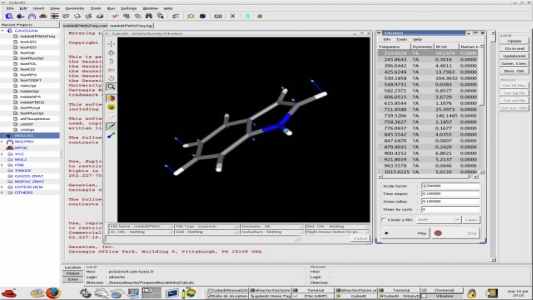
- Gabedit (gabedit.sourceforge.net, paquete gabedit): programa para 'modelado molecular' interfaz de MPQC (paquete mpqc), GAMESS-US, Gaussian, Molcas, Molpro y Q-Chem.
2 Comentarios en “Modelado molecular en Linux”
Deja un comentario

gracias por la info, esta buena. Sin embargo solo como una pequeña critica seria bueno que especifiques los tipos de métodos y los métodos con los que cuentan (MM, semi-emp, ab-initio, etc.).
De todas maneras probare algunos de ellos que no conocía ;).
la informacion me ayudo un poco, pero siento que les hace falta especificar un poco más para que se entienda un poco más
Pero esta todo bien
Gracias!!